Skuteczność testu Prenatal testDNA została potwierdzona w dużym dużym badaniu na ponad 70 000 ciążach, przeprowadzonym w jednym ośrodku.
Dane udostępnione zostały w Prenatal Diagnosis w publikacji: „Test performance and clinical utility of expanded non‐invasive prenatal test: Experience on 71,883 unselected routine cases from one single center.” (Diagn, 2024 Apr 30 doi.org/10.1002/pd.6580).
Czym wyróżnia się walidacja Prenatal testDNA?
Walidacja to badanie, które ma na celu potwierdzenie skuteczności testu prenatalnego w wykrywaniu nieprawidłowości. Oto co wyróżnia walidację Prenatal testDNA:
- Skuteczność badania została potwierdzona również dla rzadkich nieprawidłowości dzięki temu, że w walidacji brała udział duża grupa ponad 70 000 ciąż.
- Została wykonana w takich samych warunkach, w jakich na co dzień analizowane są próbki naszych pacjentek.
- Została przeprowadzona w jednym ośrodku co pozwala lepiej ocenić skuteczność testu niż wówczas, gdy dane pochodzą z różnych źródeł.
Skuteczność Prenatal testDNA
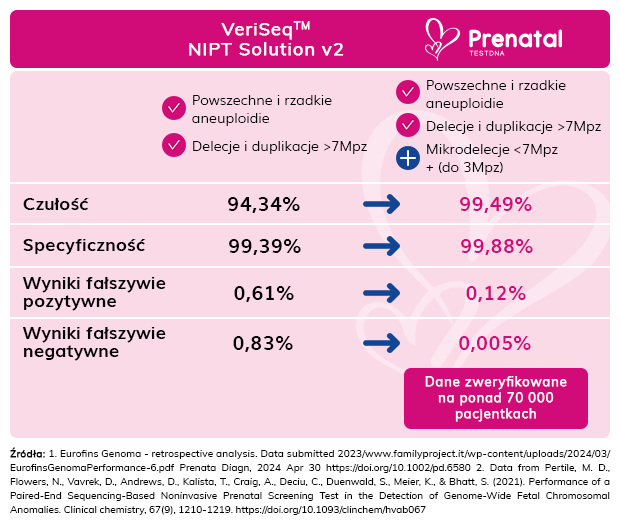
Próbki uzyskane od ponad 70 000 ciężarnych kobiet (zarówno z ciąż pojedynczych, jak i bliźniaczych) zostały przeanalizowane pod kątem obecności powszechnych trisomii, aneuploidii chromosomów płci, rzadkich aneuploidii autosomalnych, nieprawidłowości segmentowych (≥7 Mpz) i mikrodelecji (<7 Mpz). Dane zostały przetworzone przy użyciu systemu Veriseq NIPT Solution v2 w połączeniu z algorytmem opracowanym przez laboratorium.
Tabela 1. Wyniki dotyczące NIPT dla powszechnych aneuploidii, SCA i innych nieprawidłowości w 71883 ciążach
| Przypadki pozytywne
n= 1011 Całkowita obserwacja n=868 |
Trisomia 21 | Trisomia 18 | Trisomia 13 | SCA | Inne nieprawidłowości* | Ogólny wynik |
| Prawdziwie pozytywne | 437 | 93 | 37 | 156 | 58 | 781 |
| Fałszywie pozytywne | 3 | 1 | 8 | 17 | 54 | 83 |
| Prawdziwie negatywne | 71392 | 71775 | 71828 | 65598 | 46577 | 70872 |
| Fałszywie negatywne | 2 | 0 | 0 | 1 | 1 | 4 |
| Czułość
(95% CI) |
99,54% (98,36% – 99,94%) | 100% (96,11% – 100,00%) | 100% (90,51% – 100,00%) | 99,36% (96,50% – 99,98%) | 98,31% (90,91% – 99,96%) | 99,49% (98,70% – 99,86%) |
| Specyficzność
(95% CI) |
100% (99,99% – 100,00%) | 100% (99,99% – 100,00% | 99.99% (99,98% – 100,00%) | 99.97% (99,96% – 99,99%) | 99,88% (99,98% – 99,99%) | 99,88% (99,86% – 99,91%) |
| PPV (95% CI) | 99,32% (97,92% – 99,78%) | 98.94% (92,91% – 99,85% | 82.22% (69.82% – 90.24%) | 90,17% (85,08% – 93,66%) | 51,79% (45,08% – 58,42%) | 90,39% (88,36% – 92,11%) |
| NPV (95% CI) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 99,99% (99,99% -100,00%) |
CI: przedziały ufności; SCA: aneuploidie chromosomów płci. Pozytywne przypadki bez obserwacji wykluczone z liczby pozytywnych w Tabeli 1 (nr): T21 (49); T18 (14); T13 (10); SCA (36); inne nieprawidłowości (34). *Uwzględniono rzadkie aneuploidie autosomalne, nieprawidłowości segmentalne i mikrodelecje.
Tabela 2. Wyniki odnoszące się do NIPT dla aneuploidii chromosomów płciowych
| Aneuploidie chromosomów płciowych | X0 | XXX | XXY | XYY |
| Prawdziwie pozytywne | 52 | 27 | 51 | 26 |
| Fałszywie pozytywne | 13 | 0 | 3 | 1 |
| Prawdziwie negatywne | 65724 | 65775 | 65747 | 65776 |
| Fałszywie negatywne | 1 | 0 | 0 | 0 |
| Czułość
(95% CI) |
98,11% (89,93% – 99,95%) | 100% (87,23% – 100,00%) | 100% (93,02% – 100,00%) | 100% (86.77% – 100,00%) |
| Specyficzność
(95% CI) |
99,98% (99,97% – 99,99%) | 100% (99,99%- 100,00%) | 99,99% (99,99% – 100,00%) | 99.99% (99,99% – 100,00%) |
| PPV (95% CI) | 80% (69,88% – 87,34%) | 100% (99,99% – 100,00%) | 94,44% (84,57% – 98,14%) | 96.3% (78,55% – 99,46%) |
| NPV (95% CI) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) |
CI: przedziały ufności; SCA: aneuploidie chromosomów płci. Pozytywne przypadki bez obserwacji wyłączone z liczby pozytywnych w Tabeli 2 (nr): X0 (18); XXX (6); XXY (7); XYY (5)
Tabela 3. Wydajność związana z NIPT dla rzadkich aneuploidii autosomalnych, nieprawidłowości segmentalnych i mikrodelecji
| Inne nieprawidłowości | RAA | Nieprawidłowości segmentowe
(>7 pz) |
Mikrodelecje*
(nieprawidłowości segmentowe <7 Mpz) |
| Prawdziwie pozytywne | 33 | 20 | 5 |
| Fałszywie pozytywne | 36 | 16 | 2 |
| Prawdziwie negatywne | 46630 | 46681 | 28743 |
| Fałszywie negatywne | 0 | 0 | 1 |
| Czułość
(95% CI) |
100% (89,42% – 100,00%) | 100% (83,16% – 100,00%) | 83,33% (35,88% – 99,58%) |
| Specyficzność
(95% CI) |
99,92% (99,89% – 99,95%) | 99,97% (99,96%- 99,99%) | 99,99% (99,99% – 100,00%) |
| PPV (95% CI) | 47,83% (39,81% – 55,96%) | 55,56% (43,37%- 67,11%) | 71,43% (37,40% – 91,27%) |
| NPV (95% CI) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) | 100% (99,99% – 100,00%) |
CI: Przedziały ufności; RAA: Rzadkie nieprawidłowości chromosomalne. Pozytywne przypadki bez obserwacji wyłączone z liczby pozytywnych w Tabeli 3 (nr): RAA (25); Nieprawidłowości segmentalne >7Mb (7); Mikrodelecje (2). *Zbadane zespoły mikrodelecji: zespół DiGeorge’a, zespół Cri-du-chat, zespół Pradera-Williego, zespół Angelmana, zespół delecji 1p36, zespół Wolfa-Hirschhorna, zespół Jacobsena, zespół Langera-Jeidiona i zespół Smitha-Magenisa.
Źródła: Diagn, 2024 Apr 30 doi.org/10.1002/pd.6580, Eurofins Genoma retrospective analysis. Data submitted in 2023 https://www.familyproject.it/wp-content/uploads/2024/03/EurofinsGenomaPerformance-6.pdf